GeneMapper1: มี peak แต่มันไม่ขึ้น label ให้.....ทำไงดี?
วันก่อนจินตนาเรียกให้ดูตัวอย่างตรวจหนึ่งที่ทำด้วยน้ำยา YFiler แล้ววิเคราะห์ใน genemapper v 4.2 ID พบว่าที่ตำแหน่ง DYS392 มี peak ชัดเจน แต่โปรแกรมไม่แสดงค่าอัลลีล ดังภาพข้างล่างนี้ครับ

ปัญหานี้เกิดจาก ในการอ่าน peak ของโปรแกรม จะมีการกำหนดไว้ว่าลักษณะของ peak ต้องมีความกว้างของ peak ที่บริเวณครึ่งหนึ่งของความสูง ไว้ ไม่เกิน 3 datapoint ดังนั้นหากไปเจอ peak ที่มีฐานค่อนข้างกว้าง โปรแกรมก็จะไม่คิดว่าเป็น peak ครับ นั่นแหละเป็นปัญหา ทีนี้หากไม่ต้องการ run ตัวอย่างตรวจนี้ใหม่ ก็ต้องเปลี่ยนการวิเคราะห์ในโปรแกรม ให้ตั้งค่าการอ่าน peak ใหม่ครับ ให้ตั้งค่าความกว้างของค่าครึ่งหนึ่งของความสูง ไว้มากกว่า 3 datapoint เช่น ตั้งเป็น 4 หรือ 5 หรือ 6 datapoint เป็นต้น ปัญหานี้ก็จะหมดไปครับ
มาลองดูวิธีการตั้งค่านะครับ เริ่มจากไปที่ genemapper manager แล้วเลือก tab Analysis Methods จากนั้นเลือกชื่อของ Analysis method ที่เราใช้ ซึ่งในที่นี้ใช้ ClassicYSTR จากนั้นกดปุ่ม open ข้างล่างครับ อ้อ ผมลืมบอกไปว่า Classic เป็นการวิเคราะห์แบบ classic ในขณะที่การตั้งค่าจากบริษัท จะให้เราเลือกใช้แบบ advance
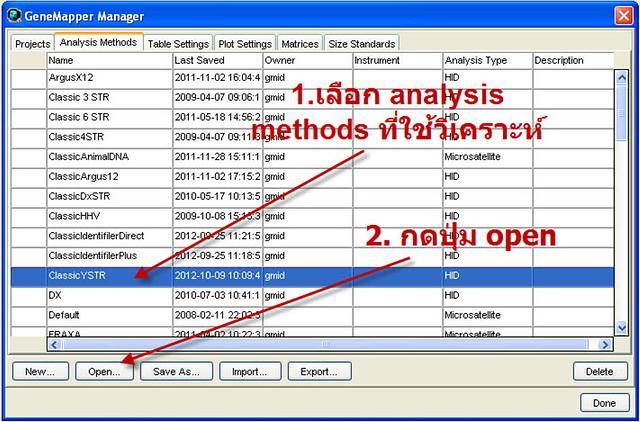
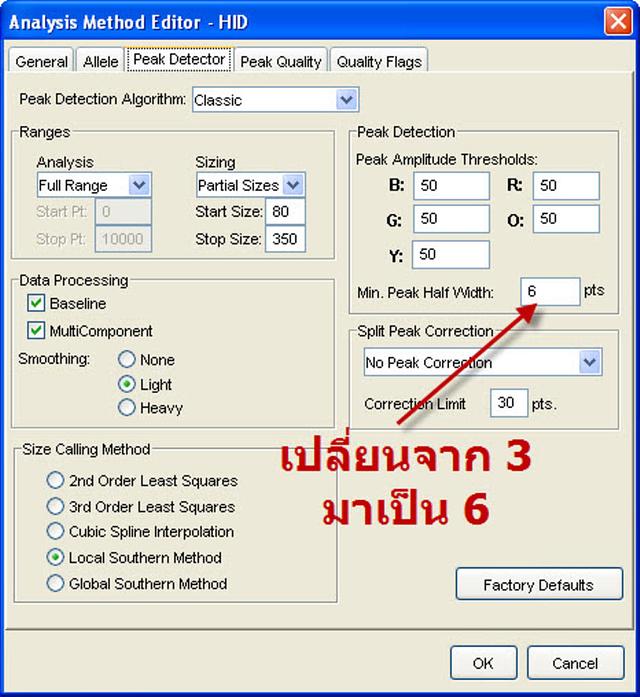
จากนั้นเลือกที่ tab Peak Detector ดูในช่อง Min Peak Half Width ค่าเริ่มต้นอยู่ที่ 2 หรือ 3 datapoint ให้ปรับเปลี่ยนค่าตัวนี้ให้มากขึ้น เช่น 4 หรือ 5 หรือ 6 datapoint ในที่นี้ผมเลือกใช้ 6 datapoint ครับ แล้วกดปุ่ม OK ออกมา แล้ว Save ข้อมูลให้เรียบร้อย
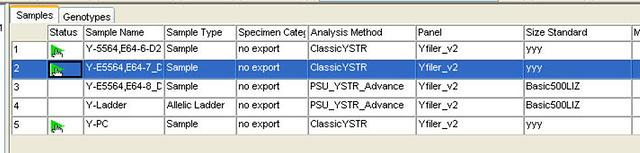
ในหน้า Samples จะมีเครื่องหมายสามเหลี่ยมสีเขียว เฉพาะในตัวอย่างที่เราเลือก Analysis method ที่แก้ไขข้อมูลไปแล้ว ให้กดปุ่ม Analysis ใหม่ครับ
จากนั้นเข้าไปดูกราฟ ก็จะมี label ให้เรียบร้อยครับ
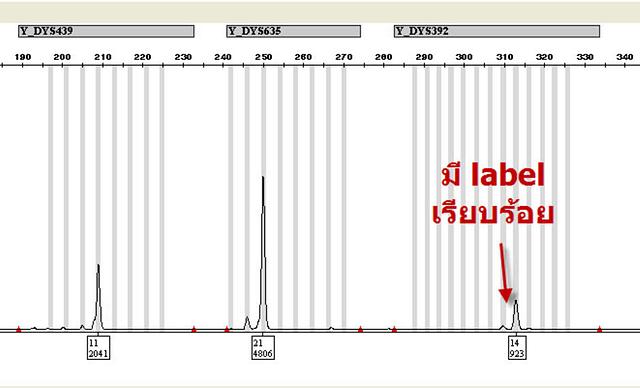
เห็นไหมว่า ไม่ต้องเสียเวลา run ใหม่ก็ได้ครับ
ความเห็น (0)
ไม่มีความเห็น