บันทึกจากแดนซากุระ 16 : เมื่อข้าม field สิ่งที่คุณเคยรู้มาลืมมันไปให้หมด แล้วเริ่มต้นเรียนรู้ใหม่
บล็อกก่อนได้มีโอกาสเล่าให้ฟังแล้วว่าจากงานที่ฝึกทำกลายมาเป็นงานวิจัยได้ มันมีหลายขั้นตอน
จะเรียกว่าเป็นอุปสรรคก็ไม่เชิง
แต่ก็ต้องการการแก้ไขปัญหาเฉพาะหน้าเหมือนกัน หลังจากที่ run PCR-RFLP
แล้วก็จะออกมาว่าเป็นกรดอมิโน Arginine หรือ Proline
ที่จริงก็แค่คำนวณ allele frequency ก็บอกเป็น percentage เท่านั้นเอง
อย่างเช่นมี Arginine เท่าไหร่ มี Proline เท่าไหร่
ก็เทียบกันออกมาเป็นเปอร์เซ็นต์ ไม่เห็นมีอะไรเลย
แต่บอกได้เลยครับว่าผิดหมด ผมก็เคยคิดอย่างนี้มาแล้ว
ผิดชนิดที่ไม่มีเค้าถูกเลยแหละ แล้วที่ถูกมันเป็นอย่างนี้ครับ
ยีนที่ควบคุมการสร้าง Arginine และ
Proline มันอยู่บน autosomal chromosome ซึ่ง chromosome
ของคนก็จะมีแขน สองข้าง ข้างหนึ่งได้จากแม่ อีกข้างได้จากพ่อ
เอามาแปะกันตรงกลางเหมือนปาท่องโก๋
แล้วเจ้ายีนที่ควบคุมมันก็จะอยู่บนแขนทั้งสองข้าง
สมมุติให้ยีนที่ควบคุม Arginine เป็น A และ Proline เป็น P
ยีนที่ควบคุมก็จะมีได้ 3 ลักษณะ ดังนี้
AA พวกนี้ได้ A
มาจากพ่อข้างหนึ่ง ได้ A มาจากแม่อีกข้างหนึ่ง (m1)
AP พวกนี้ได้ A
มาจากพ่อหรือแม่ข้างหนึ่ง และได้ P มาจากแม่หรือพ่ออีกข้างหนึ่ง
(m2)
PP พวกนี้ได้ P มาจากพ่อข้างหนึ่ง
และได้ P มาจากแม่อีกข้างหนึ่ง (m3)
เวลาคำนวณเราจะต้องแตกเจ้า AA
ออกมาเป็น 2A แล้วก็แตกเจ้า AP ออกมาเป็น 1A กับ 1P และแตก PP
ออกมาเป็น 2P ที่นี้แล้วค่อยเอามาคำนวณ จะได้ว่า
frequency (A) + frequency (P) = 1
เพราะฉะนั้น
frequencyของ A = (2m1+m2)/2*(m1+m2+m3)
และ
frequency ของ B = (m2+2m3)/2*(m1+m2+m3)
(ดูภาพด้านล่างประกอบ)
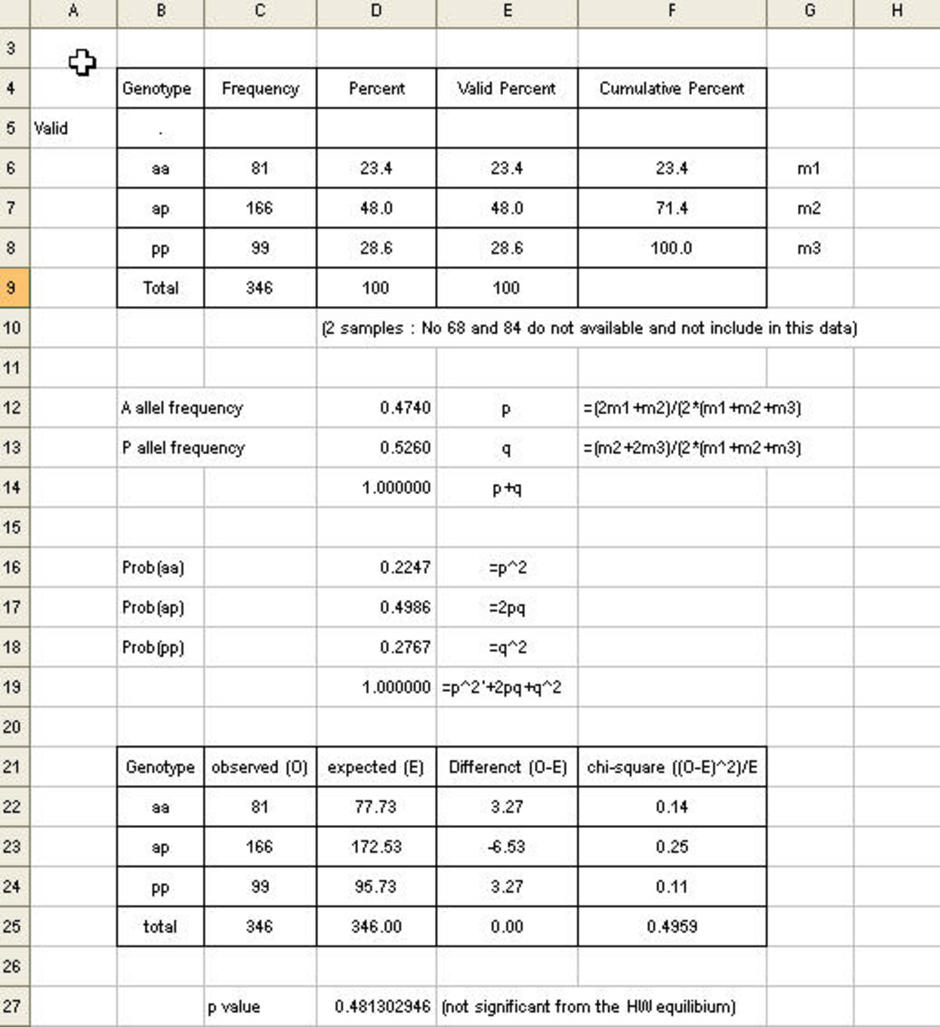
ทีนี้เราก็คำนวณ allele frequency ได้แล้ว ก็มาคำนวน Probability
ของแต่ละ genotype ก็มีสูตรดังภาพข้างบน
ส่วนที่มาของสูตรหาอ่านได้ตามหนังสือ population genetics ทั่วไปครับ
โดยค่า p ก็คือ allele frequency ของ Arginine และ q คือ allele
frequency ของ Proline
ถัดมาก็คำนวณค่า chi square
ค่าที่เรามีอยู่ก็คือค่า observe ส่วนค่า Expected
เป็นค่าที่ได้จากการคำนวณครับ โดยคำนวณจาก ค่า prob ของแต่ละ allele
คูณกับค่า Total เช่น genotype aa มีค่า prob (aa) = 0.2247
เอามาคูณกับค่า total ทั้งหมด 346 ราย รวมเป็นค่าคาดหวังว่าจะพบ
genotype aa = 77.73 เป็นต้น genotype
อื่นก็ทำลักษณะเดียวกัน
ส่วนค่า chi square ได้จากการหา
different ระหว่างค่าที่เรา observe กับค่าคาดหวัง
(ดูภาพด้านบนประกอบ) แล้วเอาค่า different
นี้ยกกำลังสองหารด้วยค่าคาดหวัง ก็จะได้ค่า chi square ของแต่ละ
genotype แล้วค่า chi square รวมก็เป็นผลรวมของค่า chi square
ที่หาได้ทั้งหมด หลังจากนั้นก็เอาไปคำนวนเป็นค่า p-value โดยใช้สูตร
chidist (ค่าผลรวมของ chi square, degree of freedom) สำหรับค่า
degree of freedom ของการหา 2 allele ก็คือ 1 ถ้าได้ค่า p-value
ออกมาต่ำกว่า 0.05 แสดงว่า กลุ่มประชากรไม่ได้อยู่ใน Hardy-Weinberge
equilibrium แต่ถ้าค่า p-value ออกมามากกว่าหรือเท่ากับ 0.05
แสดงว่ากลุ่มประชากรอยู่ใน Hardy-Winberge equilibrium ครับ
เห็นไหมว่าคำนวณไม่ยาก
เพียงแต่อย่าไปสนใจของเก่า เริ่มต้นเรียนของใหม่กันดีกว่า
คำสำคัญ (Tags): #uncategorized
หมายเลขบันทึก: 17029เขียนเมื่อ 27 กุมภาพันธ์ 2006 21:09 น. ()ความเห็น (2)
ขอบคุณมากค่ะที่เผยแพร่เรื่องดีๆ อยากรู้เรื่องนี้มานานแล้ว
mitochondria
ด้วยความยินดีครับอาจารย์
รู้สึกดีจังเลยที่ความรู้ที่ร่ำเรียนมาเป็นประโยชน์กับผู้อื่นบ้าง