How to set a new panel of fluorescent based STR-PCR ? 3: Set a new panel in Gene Mapper ID software
คราวนี้มาเริ่มของจริงกันล่ะ เราจะมาว่ากันด้วยเรื่อง การ set new panel ในโปรแกรม Gene Mapper ID นั่นคือการตั้งชื่อ panel การใส่ชื่อ marker และ การ set bin ซึ่งทั้งหมดนี้ ผมทำอยู่บนโปรแกรม Gene Mapper ID 3.2.1 ดังนั้น ท่านที่ใช้ GeneMapper รุ่นที่แตกต่างจากนี้ อาจมีหน้าตาของหน้าจอแตกต่างกันบ้างเล็กน้อย แต่ในหลักการแล้วยังเหมือนเดิมครับ
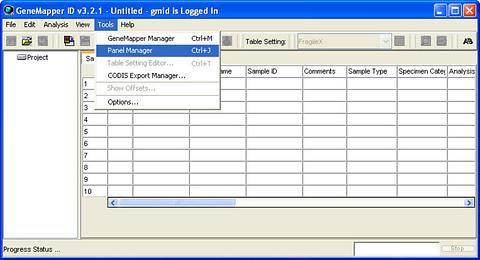
ขั้นแรก เมื่อเปิดโปรแกรม Gene Mapper รุ่น 3.2.1 ขึ้นมาแล้ว ให้ไปที่เมนู Tools แล้วเลือก Panel Manager ดังภาพ
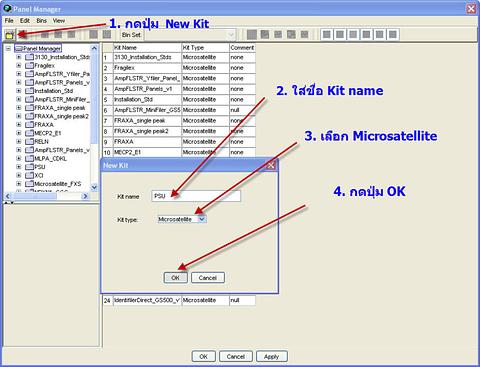
จากนั้น ก็เลือก folder ที่จะเก็บไฟล์ panel นี้ ถ้ายังไม่มี ก็ให้เลือกเมนู File แล้วเลือก New Kit จากนั้นพิมพ์ชื่อ folder (Kit Name) ที่ต้องการ ในที่นี้ ผมพิมพ์ชื่อว่า PSU แล้วกดปุ่ม OK
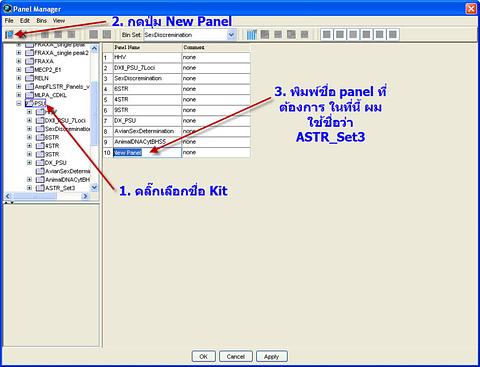
จากนั้นคลิ๊กเลือกที่ ชื่อ Kit Name ที่ใส่ไว้ นั่นคือ PSU สำหรับท่านที่ใส่ชื่ออะไรไว้ ก็ให้เลือกที่ชื่อนั้นครับ แล้วกดปุ่ม New Panel แล้วพิมพ์ชื่อ panel ที่ต้องการครับ ในที่นี้ผมตั้งชื่อว่า ASTR_Set3
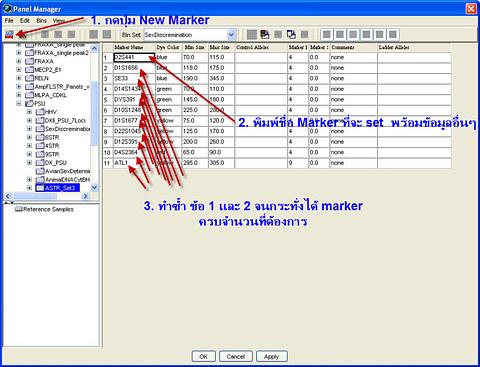
จากนั้นก็กดปุ่ม New Marker แล้วพิมพชื่อ ASTR ตำแหน่งต่างๆ ที่เราจะ set เข้าไปครับ ช่องถัดมาคือ ชื่อสีฟลูออเรสเซนต์ (ไม่มีแถบให้เลือก ต้องพิมพ์เข้าไปเอง) ในที่นี้เราจะใช้ได้แค่ 4 สี คือสี blue, green, yellow, และ red ก็ให้พิมพ์เข้าไปเลยครับ ระวังเรื่องการพิมพ์ผิดสักหน่อย แล้วให้ใช้ตัวอักษรพิมพ์เล็กทั้งหมดครับ
ช่องถัดมาคือ Min Size และ Max Size เป็นช่องสำหรับใส่ขอบเขต ของ PCR product ของ autosomal STR แต่ละตำแหน่งครับ อย่างเช่น D2S441 มีขนาด PCR product อยู่ในช่วง 70-115 bp เราก็พิมพ์ค่า 70 ลงไปในช่อง Min size และพิมพ์ค่า 115 ลงไปในช่อง Max Size
ช่องถัดมาคือช่อง control alleles ให้เราค้างไว้ก่อน ยังไม่ต้องพิมพ์อะไรเข้าไป
ช่องถัดมาคือช่อง marker repeat คือค่า core base ของ autosomal STR ตำแหน่งนั้นๆ หมายถึง base แกนกลาง ของตำแหน่งนี้ ว่ามีทั้งหมดกี่เบส ส่วนใหญ่ที่เราทำ autosomal STR มักเป็น 4 เบส ครับ แต่ถ้าตำแหน่งไหนเป็น 5 เบส ก็ให้พิมพ์ตัวเลข 5 เข้าไปครับ สำหรับตำแหน่งที่ไม่เป็น STR เช่น amelogenin ก็ให้พิมพ์เลข 9 เข้าไปแทนครับ ยกตัวอย่างในที่นี้ ผมเพิ่มตำแหน่ง ATL1 ลงไปด้วย ตำแหน่ง ATL1 เป็นตำแหน่ง gene ที่อยู่บนโครโมโซม X ครับ ไม่เป็น microsatellite เป็นตำแหน่งที่เอาไว้ตรวจสอบว่ามี gene ATL1 อยู่หรือไม่ ถ้ามี ก็แสดงว่า มี X-chromosome อยู่ด้วย ดังนั้นตำแหน่ง ATL1 ผมจึงพิมพ์เลข 9 เข้าไปในช่อง marker repeat
ส่วนตำแหน่งที่เหลือ ให้ว่างไว้ก่อนครับ ยังไม่ต้องไปยุ่งกับมันก่อน
ทำซ้ำขั้นตอนนี้ จนกระทั่งเราใส่ marker ได้ครบตามจำนวนที่ต้องการ จากนั้นก็กดปุ่ม OK ครับ
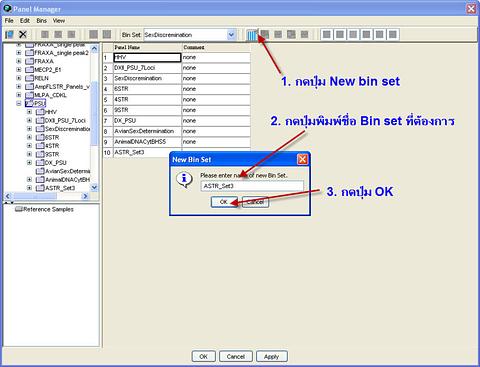
จบเรื่องการจัดการ panel ทีนี้ก็มาจัดการเรื่อง bin set ครับ เรื่องนี้เป็นการบอกให้โปรแกรมรู้ว่า ในแต่ละตำแหน่งจะมี allele อะไร ขนาดกี่ซ้ำ แต่ละซ้ำมีขนาดกี่เบส ครับ เริ่มต้นก็เราเพียงกำหนดว่า เราจะจัดตั้ง bin set ใหม่ โดยกดปุ่ม new bin set ดังภาพข้างล่างนี้ครับ จากนั้นจะมีหน้าต่างขึ้นมา ก็พิมพชื่อ bin set ที่เราต้องการ อย่างเช่นในที่นี้ ผมตั้งชื่อว่า ASTR_Set3 แล้วก็ก็ปุ่ม OK ครับ ท่านสามารถตั้งชื่อ bin set เป็นชื่ออะไรก็ได้ครับ เพียงแต่ตั้งชื่ออะไรไว้ ก็จำไว้ด้วยก็แล้วกันครับ
เรื่อง bin set เป็นเรื่องที่ต้องจัดการต่อครับ แต่ยังไม่ใช่ตอนนี้ เพราะเรายังไม่รู้ว่า ในแต่ละตำแหน่งของเรา ที่เราจะต้องทำ pool ladder นั้น จะมีกี่ allele และแต่ละ allele จะมีขนาดเท่าใด เพราะฉะนั้น ให้ว่างไว้ก่อนครับ เรื่องนี้ยังไม่ต้องทำ
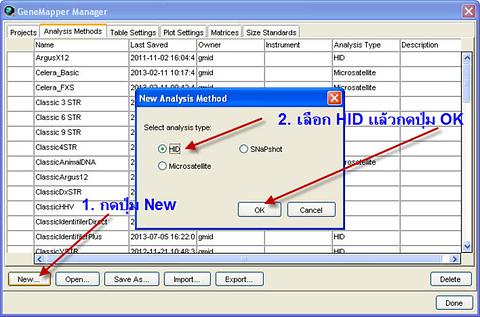
เรื่องต่อไปที่ต้องจัดการคือตั้งค่า Analysis Methods ใหม่ คราวนี้ให้กลับไปที่เมนูหลัก หน้าแรกที่เปิดโปรแกรมเลย แล้วเลือกเมนู Tools เลือก GeneMapper Manager จะได้หน้าต่างดังภาพข้างล่างครับ จากนั้นกดปุ่ม New โปรแกรมจะแสดงหน้าต่าง New Analysis Method ให้เลือก HID แล้วกดปุ่ม OK ครับ
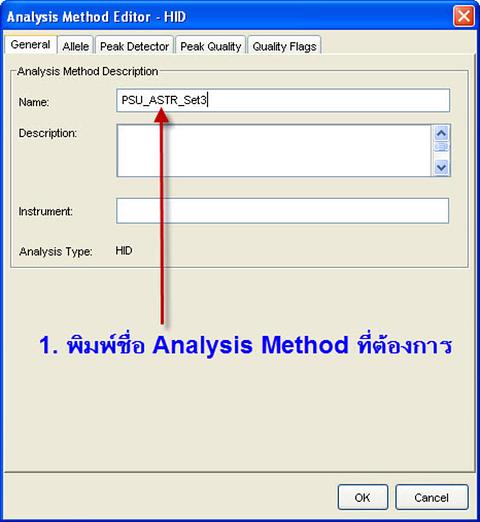
หน้าต่าง Analysis Method Editor จะมีหลาย tab ครับ หน้าแรกเป็น General ให้เราใส่ชื่อ Analysis Method ครับ ในที่นี้ ผมตั้งชื่อว่า PSU-ASTR_Set3
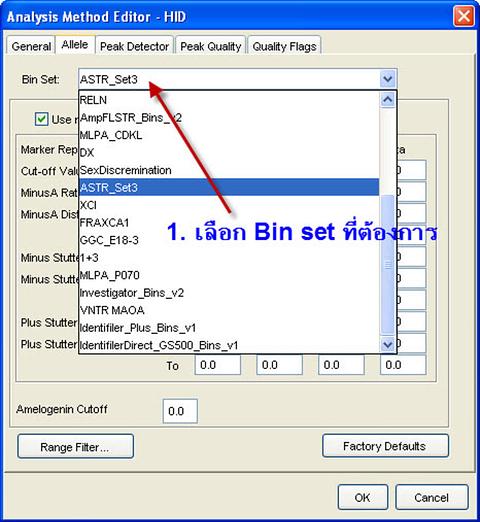
แล้วกด tab allele ครับ จากนั้นเลือก Bin set โดยเลือกจาก drop down list ครับ ยังจำได้หรือปล่าว ว่าตั้งชื่อ Bin set ไว้ว่าอย่างไร
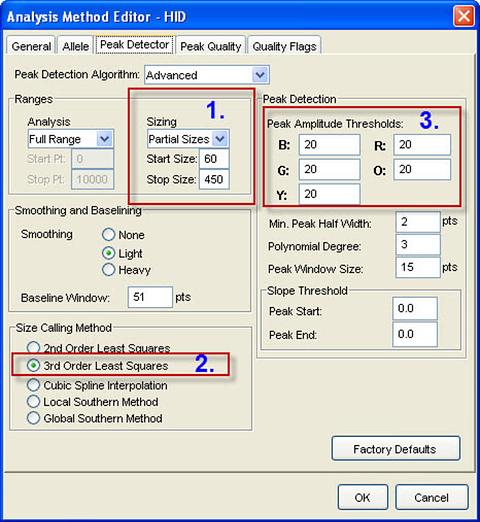
จากนั้นเลือก tab ที่ชื่อว่า Peak Detector ในหน้านี้ มีเรื่องที่ต้อง set อยู่อย่างน้อย 3 เรื่องครับ
ก่อนที่จะขึ้นเรื่องที่ 1 ให้ชำเลืองดูในช่อง Peak Detection Algorithm ก่อนว่าเป็น Advanced หรือเปล่า ช่องนี้ มีให้เลือก แค่สองตัว คือ Advanced กับ Classic เรื่อง algorithm นี้ เอาไว้ค่อยว่ากันวันหลังครับ วันนี้ให้เลือกเป็น Advanced ไว้ก่อน ซึ่งค่านี้น่าจะเป็น ค่า default ของโปรแกรมครับ
เรื่องแรกคือ ค่า Ranges หรือช่วง ที่จะให้เครื่องวัดค่าคับ ให้เราเลือก Sizing เป็น Partial Sizes แล้วใส่ค่าเริ่มต้นไว้ที่ 60 และ สิ้นสุดที่ 450 ค่านี้จะมีหน่วยเป็น bp ครับคือจะให้เครื่องอ่านค่าตั้งแต่กี่เบสไปจนสิ้นสุดที่กี่เบส
เรื่องที่ 2 คือเรื่อง Size calling Method ซึ่งผมเคยเขียนเป็นบันทึกไว้ก่อนหน้านี้แล้ว คงไม่ต้องอธิบายซ้ำอีก ค่า default จะเป็นค่า Local Southern Method แต่คราวนี้ เราไม่ได้ใช้ค่านี้ครับ เพราะ autosomal STR ที่เรา set เพิ่มคราวนี้ เป็น mini STR หลายตัว โดยมีขนาด เริ่มต้น ตั้งแต่ 67 bp ดังนั้น เราจึงไม่สามารถใช้ calling method แบบ Local Southern Method ได้ ลักษณะของ autosomal STR ที่เป็น mini STR คือมีค่าที่ต่ำกว่า 100 bp นั้น ให้เราเลือก calling method แบบ 3rd Order Least Squares ครับ
แล้วเรื่องที่ 3 คือ peak detection คือการกำหนดค่า peak amplitude thresholds ของแต่ละสีว่าต้องมีค่าไม่น้อยกว่าเท่าไหร่ จึงจะอ่านค่า peak ให้ครับ ค่า default กำหนดไว้ที่ 50 rfu แต่โดยทั่วไปผมมักตั้งต่ำกว่านั้น ในที่นี้ผมตั้งไว้ที่ 20 rfu ซึ่งโปรแกรมจะอ่านค่า peak ทุก peak ที่มีค่าสูงกว่า 20 rfu ครับ ประโยชน์เรื่องนี้คือ สีส้ม ซึ่งเป็นสีของ LIZ ที่ใช้เป็น internal size standard การที่ผมตั้งให้อ่านค่าได้ต่ำ จะทำให้ผมสามารถเจือจาง Liz ได้มากขึ้นครับ ส่วนสีอื่นๆ ก็จะช่วยในกรณีที่เราทำ PCR โดยใส่ ดีเอ็นเอปริมาณไม่มากนัก ทำให้มี peak ไม่สูง โปรแกรมก็อา่น peak ได้ครับ แต่ข้อดีนี้ ก็ต้องแลกมาด้วยข้อเสีย คือกรณีที่ใส่ดีเอ็นเอเข้าไปในปฏิกิริยาค่อนข้างมาก โปรแกรมจะอ่าน peak เตี้ยๆ ได้เป็นตับเลยครับ ก็ต้องมาเสียเวลาลบ peak ที่เป็น non specific ออกไป ทำให้เสียเวลามากขึ้น อย่างว่าครับ มีได้ ก็ต้องมีเสีย แล้วแต่จะเลือกครับ ค่านี้ถ้าไม่คิดอะไรมาก ก็ให้เลือกที่ระดับ 50 rfu ไว้ครับ น่าจะเป็นค่า ที่ optimize ไว้ได้ดีอยู่แล้ว อย่ามาเลือกตามผมเลย จากนั้น ก็กดปุ่ม OK
นั่นคือการ set panel ใน Gene Mapper ครับ ซึ่งเป็นการ set เพียงแค่เริ่มต้น เพื่อให้สามารถใช้งานในการสั่งทำการทดสอบได้เท่านั้น เรายังไม่ได้ set ให้โปรแกรมสามารถอ่านค่าจำนวนซ้ำ ออกมาเลยครับ เพราะตอนนี้เรายังไม่มีข้อมูลเรื่องนั้นครับ ก็เลยกระโดดข้ามไปก่อน
เรื่องนี้ยังไม่จบครับ ถัดจากนี้ไป ก็ต้องไปตั้ง result group ในโปรแกรม Data collection ซึ่งเป็นโปรแกรมที่สั่งให้เครื่อง sequencer ทำงานและเก็บข้อมูลครับ
To Be Continue.....ครับ
ความเห็น (2)
ขอบคุณมากครับ ![]() สำหรับ ชมพู่เมืองเพชร
สำหรับ ชมพู่เมืองเพชร
แหม ถ้าเปลี่ยนจากภาพ ไปเป็นของจริงล่ะก้อ.....สุดยอดเลยครับ
อิ อิ